BrestCancerデータセットをCNNで分類する

構造化データを畳み込みニューラルネットワーク(CNN)で分析することを考えます。BrestCancerデータセットはScikit-learnに用意されている、乳がんが良性か悪性かの2種類を分類する典型的な構造化データです。サンプル数569、データの次元30の典型的な構造化データです。
目次
なぜ畳み込みニューラルネットワーク(CNN)なのか
CNNは画像分類など非構造化データに対して使われる例が多いですが、本質的にはBrestCancerデータセットのような構造化データに対しても使えます。なぜ畳み込みなのかと言われると、例えば「a, b, c」という3つの次元に対して、「1/3, 1/3, 1/3」をカーネルとした畳みこみを行ったとしてましょう。この結果は、
$$ (a+b+c)/3$$
であり、これは結果的には移動平均を取っているのと同じになります。また、「a, b」という2つの次元に対して「1, -1」という畳込みを行うと、
$$ a-b$$
という差分を取っているのと同じです。つまり、1次元の畳込み処理により、移動平均、差分といった主に時系列解析で使われる手法を内生的に表現でき、どのような処理を行わせるかは訓練を通じてモデルに選ばせるということができます。もう少しわかりやすく言うと、「画像のように系列間に相関がある場合は、パラメーター間の相関に着目して訓練が進むCNNもやってみる価値があるよ」ということです。
では30次元の構造化データをどのようにCNNに落とし込むのでしょうか。図で表現してみます。
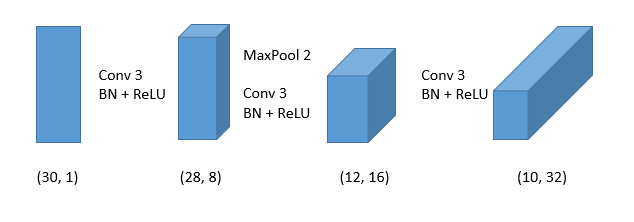
入力を「データ数×30×1」にすると1次元の畳み込みの問題に落とし込めます。あとはConv+BatchNorm+ReLUといういつものパターンでできます。
ではやってみましょう。最初にデータをfeature-wiseで標準化しておきます。
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from keras.layers import Conv1D, BatchNormalization, Activation, MaxPool1D, Flatten, Dense, Input
from keras.models import Model
from keras.optimizers import Adam
from keras.utils import to_categorical
import numpy as np
import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True)
X = X.reshape(-1, 30, 1)
y = to_categorical(y.reshape(-1, 1))
# featurewiseな標準化
X = (X - np.mean(X, axis=0)) / np.std(X, axis=0)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=0)
def create_single_module(input_tensor, output_channel):
x = Conv1D(output_channel, kernel_size=3)(input_tensor)
x = BatchNormalization()(x)
return Activation("relu")(x)
input = Input(shape=(30, 1)) # Brest-cancerは30次元
x = create_single_module(input, 8) # 30->28 dim
x = MaxPool1D(2)(x) # 28 -> 14dim
x = create_single_module(x, 16) # 14 -> 12
x = create_single_module(x, 16) # 12 -> 10
x = MaxPool1D(2)(x) # 10 -> 5
x = create_single_module(x, 32)
x = Flatten()(x)
x = Dense(2, activation="softmax")(x)
model = Model(input, x)
model.compile(Adam(lr=1e-3), loss="binary_crossentropy", metrics=["acc"])
history = model.fit(X_train, y_train, batch_size=32, epochs=100, validation_data=(X_test, y_test)).history
max_val_acc = max(history["val_acc"])
plt.plot(np.arange(100)+1, history["val_acc"])
plt.ylim((0.9, 1))
plt.title(f"BrestCancer in CNN / max val_acc = {max_val_acc:.3}")
plt.show()
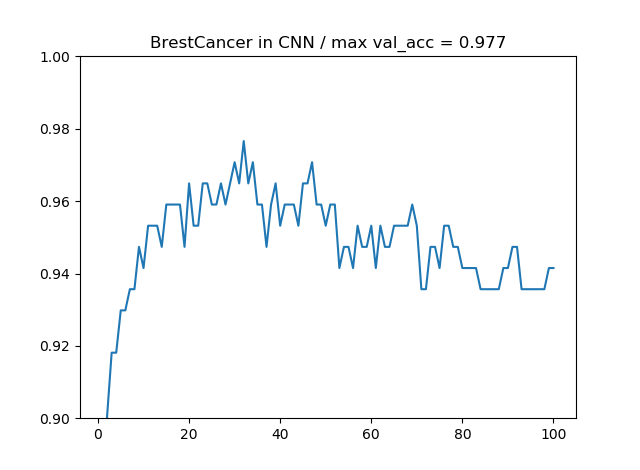
CNNを使った場合は最大精度が97.7%となりました。
多層パーセプトロンでもやってみる
通常よく出てくるのはこちらです。多層パーセプトロンはすぐオーバーフィッティングするのでL2正則化を入れながらやってみます。
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from keras.layers import Dense, Input, Activation, BatchNormalization
from keras.models import Model
from keras.optimizers import Adam
from keras.regularizers import l2
from keras.utils import to_categorical
import numpy as np
import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True)
y = to_categorical(y.reshape(-1, 1))
# featurewiseな標準化
X = (X - np.mean(X, axis=0)) / np.std(X, axis=0)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=0)
def create_single_dnn_module(input_tensor, hidden_units, l2_param):
x = Dense(hidden_units, kernel_regularizer=l2(l2_param))(input_tensor)
x = BatchNormalization()(x)
x = Activation("relu")(x)
return x
input = Input(shape=(30,))
x = create_single_dnn_module(input, 25, 3e-2)
x = create_single_dnn_module(x, 20, 2e-2)
x = create_single_dnn_module(x, 12, 1.5e-2)
x = create_single_dnn_module(x, 8, 1e-2)
x = create_single_dnn_module(x, 4, 5e-3)
x = Dense(2, activation="softmax")(x)
model = Model(input, x)
model.compile(Adam(lr=1e-3), loss="binary_crossentropy", metrics=["acc"])
history = model.fit(X_train, y_train, batch_size=32, epochs=100, validation_data=(X_test, y_test)).history
max_val_acc = max(history["val_acc"])
plt.plot(np.arange(100)+1, history["val_acc"])
plt.ylim((0.9, 1))
plt.title(f"BrestCancer in DNN / max val_acc = {max_val_acc:.3}")
plt.show()
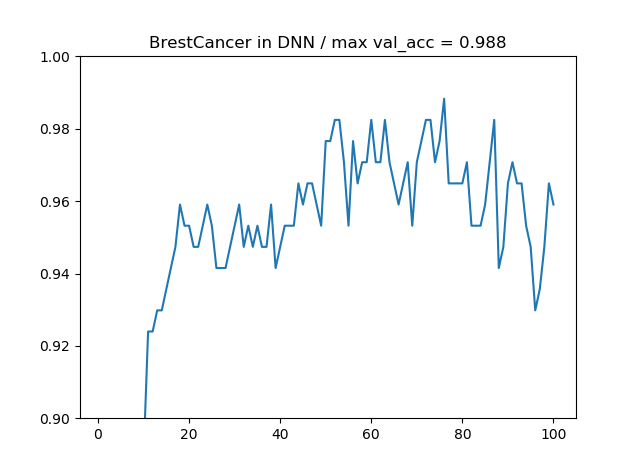
多層パーセプトロンの場合、最大精度が98.8%となりました。たまたま今回は多層パーセプトロンがうまくいきましたが、多層パーセプトロンは次元が多くなるとパラメーター数が爆発的に多くなるのでご注意ください。あとよくオーバーフィッティングします。
それサポートベクトルマシンでよくない?
というディープラーニングあるあるなツッコミがきそうなので、サポートベクトルマシン(SVM)でどれぐらいの精度が出るか見てみます。
SVMにはγとCという2つのハイパーパラメータがあるので、SklearnでざっくりGridSearchしてみます。
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import GridSearchCV
from sklearn.svm import SVC
import numpy as np
X, y = load_breast_cancer(return_X_y=True)
# featurewiseな標準化
X = (X - np.mean(X, axis=0)) / np.std(X, axis=0)
# グリッドサーチのパラメーター
params = [{"kernel":["poly"], "degree":[2,3,4,5,6],"gamma":[1e-4, 1e-2, 1],
"C":[1e-2, 1, 100, 10000]}]
clf = GridSearchCV(SVC(), params, scoring="accuracy")
clf.fit(X, y)
for mean, std, params in zip(clf.cv_results_['mean_test_score'],
clf.cv_results_['std_test_score'], clf.cv_results_['params']):
print("%0.3f (+/-%0.03f) for %r"
% (mean, std * 2, params))
結果はこちら
0.627 (+/-0.003) for {'C': 0.01, 'gamma': 0.0001, 'kernel': 'rbf'}
0.627 (+/-0.003) for {'C': 0.01, 'gamma': 0.01, 'kernel': 'rbf'}
0.627 (+/-0.003) for {'C': 0.01, 'gamma': 1, 'kernel': 'rbf'}
0.745 (+/-0.046) for {'C': 1, 'gamma': 0.0001, 'kernel': 'rbf'}
0.965 (+/-0.020) for {'C': 1, 'gamma': 0.01, 'kernel': 'rbf'}
0.631 (+/-0.007) for {'C': 1, 'gamma': 1, 'kernel': 'rbf'}
0.967 (+/-0.022) for {'C': 100, 'gamma': 0.0001, 'kernel': 'rbf'}
0.968 (+/-0.009) for {'C': 100, 'gamma': 0.01, 'kernel': 'rbf'}
0.631 (+/-0.007) for {'C': 100, 'gamma': 1, 'kernel': 'rbf'}
0.967 (+/-0.010) for {'C': 10000, 'gamma': 0.0001, 'kernel': 'rbf'}
0.956 (+/-0.013) for {'C': 10000, 'gamma': 0.01, 'kernel': 'rbf'}
0.631 (+/-0.007) for {'C': 10000, 'gamma': 1, 'kernel': 'rbf'}
SVMでは97%ぐらいの精度が出るようです(カーネルがpolyの場合は6次元まで調べてみましたが95%ぐらいでした)。つまり、BrestCancerデータセットの場合、CNNを使っても多層パーセプトロンを使っても、SVMの精度は越えるということがわかります。いくらカーネルトリックで非線形な処理しているといっても、ニューラルネットワークでは隠れ層を突っ込めば表現できる非線形さが際限なく増えますからね。
ただ、(Sklearnの)SVMには決定的な欠陥があります。それはGPUが使えないということです。今はGoogle Colabを使えば誰しも無料でGPU訓練ができるので、GPUが使えないという欠陥はかなり大きいです。この差が気になるのなら、例えばMNISTをRBFカーネルのSVMを使ってCPUで訓練させてみてください。MNISTクラスでも物凄い時間がかかりますので。
まとめ
BrestCancerデータセットを通じて、CNNでも構造化データに対してSVMを越える程度の精度が出ることを確かめられました。CNNが万能というわけではありませんが、多層パーセプトロンだけではなくCNNも構造化データに対する1つの選択肢として検討してみてくださいね。
Shikoan's ML Blogの中の人が運営しているサークル「じゅ~しぃ~すくりぷと」の本のご案内
技術書コーナー
北海道の駅巡りコーナー

